|
I
|
NTERPRETANDO EL COEFICIENTE DE CONSANGUINIDAD O ENDOGAMIA |
|
www.labordadurtx.es
>> divulgación >>
|
En el artículo “Parentesco y consanguinidad-2” de esta misma web hemos definido el concepto del coeficiente de consanguinidad, -también llamado de endogamia- y el método de cálculo para obtener su valor, ventajas o desventajas de realizarla y la importancia de seguir con una estricta política de selección. En el presente artículo vamos a relatar cómo debemos de interpretar este valor.
Definida la endogamia como el acto de aparear individuos emparentados (consanguíneos), nos debemos de preguntar cómo hemos de interpretar el valor de su coeficiente. Por otro lado sabemos que si durante muchas generaciones se aparean individuos emparentados, la proporción de pares de genes homocigotos de los descendientes aumentará ininterrumpidamente. Es decir, los descendientes se parecerán cada vez más entre sí en aspecto y comportamiento. La proporción de estos pares de genes homocigotos en un grupo interreproductivo de animales se conoce como homocigosidad, y la proporción de pares de genes heterocigotos en el grupo se denomina heterocigosidad.
El coeficiente de consanguinidad F puede variar de 0 (endogamia nula) a la unidad (endogamia total) que -como hemos dicho- mide el aumento de la proporción de homocigosidad. Pero –como es lógico- a su vez también se produce una disminución de la heterocigosidad cuyo valor es 1-F. Pero lo que realmente importa en la valoración del grado de endogamia es la tasa de la disminución de heterocigosidad -que llamaremos T-(total por generación) con que se produce. Esta tasa se obtiene tomando la raíz k de 1-F, donde k es el número de generaciones del pedigree desde que se empezó la endogamia.
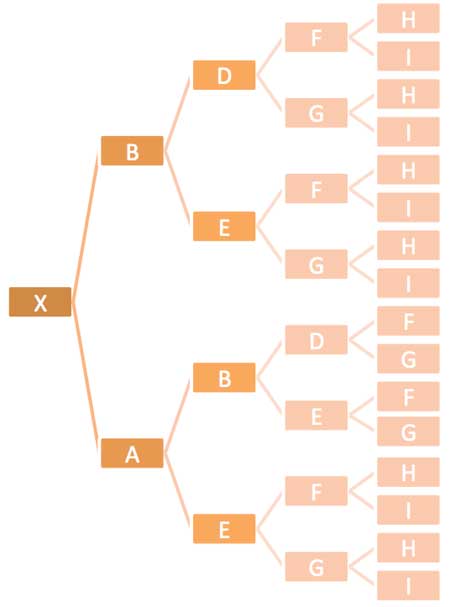
Para ilustrarlo pongamos el ejemplo de la figura 13 situado en el mencionado artículo “Parentesco y consanguinidad-2” de esta misma web para ver cómo se calcula Fx:

Primero busquemos todos los trayectos posibles de acuerdo con el método de cálculo detallado en el mencionado artículo “Parentesco y consanguinidad-2”:
X es endógamo del ejemplar H: trayecto de F hasta G: FDBXAEG = (1/2)7 = 0,0078125
X “ “ “ H: “ de G hasta F: GDBXAEF = (1/2)7 = 0,0078125
X “ “ “ I: “ de F hasta G: FDBXAEG = (1/2)7 = 0,0078125
X “ “ “ I: “ de G hasta F: GDBXAEF = (1/2)7 = 0,0078125
X “ “ “ F: “ de D hasta E: DBXAE = (1/2)5 = 0,03125
X “ “ “ G: “ de D hasta E: DBXAE = (1/2)5 = 0,03125
X “ “ “ E: “ de B hasta A: BXA = (1/2)3 = 0,125 x (1 + 0,250) = 0.15625
(Se ha añadido a E el factor (1 +0,250) por ser endógamo de los ejemplares H e I mediante dos trayectos FEG = (1/2)3 x 2 = 0,250)
X es endógamo del ejemplar B: trayecto de X hasta A: XA = (1/2)2 = 0,25 x (1 + 0,375) = 0,34375
(A su vez B es endógamo de los ejemplares siguientes:
de H: trayecto de F hasta G: FDBEG = (1/2)5 = 0,03125
de H: " " G “ F: GDBEF: (1/2)5 = 0,03125
de I: " “ F " G: FDBEG: (1/2)5 = 0,03125
de I: " “ G " F: GDBEF: (1/2)5 = 0.03125
de F: " “ D " E: DBE : (1/2)3 = 0,125
de G: " “ D " E: DBE : (1/2)3 = 0,125
_________
Total de la endogàmia de B: FB = 0,375 ) por eso se ha añadido a B el factor (1 +0,375)
Sumando los resultados de todos los trayectos nos da: 0,59375
Entonces:
Fx = 0,59375 = 59,37%, un índice de consanguinidad de moderada a alta, pero no nos podemos quedar solamente con este índice. De ahí viene la necesidad de calcular el valor de la tasa T que hemos descrito anteriormente:
Por lo tanto, previamente hemos de calcularla disminución de la heterocigosidad que se ha producido después de los cruces, o sea 1 - Fx = 1 - 0,59375 = 0,40625 que equivale a la heterocigosidad actual en relación a la existente antes de que empezase la endogamia.
Entonces, como que en el ejemplo de la figura 13 vemos que el individuo X es el resultado de cuatro generaciones de endogamia, ya estamos en condiciones de calcular T:
T = ![]()
T = ![]()
T = 0,7984
O sea, que aunque el índice de consanguinidad es medio-alto, vemos que la tasa de disminución proporcional de la heterocigosidad por cada generación no es excesiva. Pero es importantísimo e imprescindible que los individuos que se usen para realizar los cruces endogámicos sean de una calidad excepcional.
En este sentido, la aportación a la consanguinidad de los antepasados mostrados en este ejemplo puede valorarse de la siguiente manera: la mayor aportación pertenece al individuo B; en primer lugar porque se halla a una y dos generaciones de distancia de X, y en segundo lugar por ser endógamo él mismo. También es importante la aportación de E que está a dos y tres generaciones de X. Las aportaciones de F y G son menos importantes porque se encuentran a 3 y 4 generaciones de X. Por último, las aportaciones de H y I son insignificantes al hallarse a 4 generaciones de X.
Al usarse B y -también E pero en menor medida-, para que lleven el peso de estos cruces endogámicos, ha sido porque ambos son de una calidad excepcional. Consecuentemente, X podrá considerarse un buen reproductor a causa de la aportación genética que recibe de ellos, ya que entre los dos suman un coeficiente de 0,5.
Aunque parezcan difíciles, los cálculos en la práctica no lo son si se procede metódicamente. El coeficiente F y la tasa T son la mejor medida posible de la endogamia cuyos valores nos permiten llevar el control genético de las razas. De hecho en nuestro caso, en la Borda d’Urtx, disponemos de un programa informático propio que nos calcula automáticamente estos valores además de detallarnos los valores parciales y los nombres de los perros que los aportan y es una herramienta muy valiosa que usamos a la hora de programar los cruces.
Joan Ferrer
Marzo 2015
Bibliografía:
Genética para Criadores de Perros. Roy Robinson.
|
JOAN FERRER i SIRVENT
LA BORDA D'URTX 17538 URTX (Girona) CATALUNYA Telf: +34-629-613399 e-mail: joanferrer@labordadurtx.org |